
本文主要使用到 LLM(大语言模型)和 RAG(搜索生成增强)两种技术。
综合考虑模型消耗的资源和模型能力,选择的是开源的 LLM —— Mistral 7B。选择 PubMed 文章作为我们的知识库,独立评估 Mistral 7B 的有效性以及与 RAG 结合使用时的有效性。目标是确定将检索步骤与 Mitral 7B 集成是否可以在医学领域产生更准确、更可靠的答案。
这种比较对于了解医疗专业人员和研究人员未来如何利用先进的人工智能工具进行高效的信息检索和决策非常重要。
硬件配置
- 节点:1
- 每节点卡数:1
- GPU型号:A6000
- GPU 内存:48G
学习内容
- 了解检索增强生成 (RAG) 与大型语言模型 (LLM) 协作带来的好处
- 学习如何将外部知识数据库 PubMed 与预训练的 LLM (Mistral 7B )相结合,以提高医学答案的准确性
- 了解如何使用 LangChain 和 FAISS 等工具构建自定义 RAG 系统
数据收集
PubMed 是一个免费搜索引擎,主要提供生命科学和生物医学主题的内容。数据保存在 JSON 文件中,结构如下:
- 文章标题
- 出版日期
- 文章摘要
本文中,前两个元素用作元数据,而第三个元素是用于我们的 RAG 系统的内容数据。用于下载数据的 Python 脚本可在此 GitHub 存储库中找到。
RAG
下图是 RAG 管道的概述:
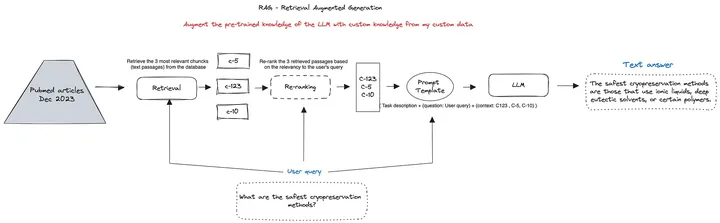
以下是 RAG 工作流程的步骤说明:
1- 自定义数据库:在我们的例子中,它对应于 2023 年 12 月的医学文章切成块并存储到自定义向量数据库中。
2-检索:当用户进行查询时,系统基于用户查询和索引块之间的相似性搜索,以文本格式从数据库中检索k个最相关的块。这些块表示为“c-5”、“c-123”和“c-10”。
3-重排:这是 RAG 系统中的一个可选步骤,通常用于确保在 LLM 提示词开头考虑最相关的块。
4- 提示词模板:提示词模板 LLM 的输入格式。此模板包括任务描述、用户的问题以及检索到的上下文。
5- LLM:格式化的提示被输入到 LLM 中,例如 Mistral 7B,它根据提供的信息生成文本答案。
6- 文本答案:最终输出是一个文本答案,它使用最相关的信息来响应用户的查询。
该工作流程演示了 RAG 系统如何利用来自目标数据库的自定义数据来增强 LLM 的预训练知识,从而为用户查询提供精确的答案。
RAG 实现
首先,安装相关的库:
pip install langchain faiss jq
LangChain 是一个旨在创建 RAG 管道的框架。Faiss 是用于组织和访问 RAG 系统所需的医疗信息的向量数据库。Sentence-transformers 是一个文本嵌入的框架。最后,jq 是一个命令行 JSON 处理器。
然后,我们使用 LangChain 的 JSONLoader 来加载我们的文档。请注意,我们还定义了一个元数据函数来提取源文档的信息。
rom langchain.document_loaders import JSONLoader
# Define the metadata extraction function.
def metadata_func(record: dict, metadata: dict) -> dict:
metadata[“year”] = record.get(“pub_date”).get(‘year’)
metadata[“month”] = record.get(“pub_date”).get(‘month’)
metadata[“day”] = record.get(“pub_date”).get(‘day’)
metadata[“title”] = record.get(“article_title”)
return metadata
loader = JSONLoader(
file_path=’./data/pubmed_article_december-2023.json’,
jq_schema=’.[]’,
content_key=’article_abstract’,
metadata_func=metadata_func)
data = loader.load()
我们检查一下加载文章的数目:
print(f”{len(data)} pubmed articles are loaded!”)
8267 pubmed articles are loaded!
知识库索引
加载 PubMed 文章后,我们需要将它们切分成更小的文本段落。对 RAG 的文档进行分块非常重要,因为它有助于将大型文本切分为可管理的部分,从而确保在检索相关信息时进行更高效的处理和更高的准确性。
此外,由于预训练 LLM 的上下文长度限制,对文档进行分块也很重要。此外,较大的输入提示会增加幻觉的风险并产生不相关的答案。在我们的测试中,块大小(chunk size)为 128, 块重叠(chunk overlap)为 50。
PS:块大小和重叠两个参数会影响答案的质量。因此,应根据数据的具体情况确定它们的值。
from langchain.text_splitter import TokenTextSplitter,CharacterTextSplitter
text_splitter = TokenTextSplitter(chunk_size=128, chunk_overlap=50)
chunks = text_splitter.split_documents(data)
接下来,我们将设置嵌入函数,它将每个文本块映射成向量。我们选择了 e5-large-unsupervised 模型,它是为通用文本嵌入而设计的。原始文本块及其相应的嵌入向量以键值对的格式存储在向量数据库中。
rom langchain.embeddings import HuggingFaceEmbeddings
modelPath = “intfloat/e5-large-unsupervised”
embeddings = HuggingFaceEmbeddings(
model_name = modelPath,
model_kwargs = {‘device’:’cuda’},
encode_kwargs={‘normalize_embeddings’:False}
# Using faiss index
from langchain.vectorstores import FAISS
db = FAISS.from_documents(chunks, embeddings)
使用相同的嵌入函数,将用户的查询编码到与存储块相同的嵌入空间中。随后,执行相似性搜索以检索与用户查询相关的前 k 个块。
现在,让我们执行一个示例查询来评估数据库的性能:
query = “What is the most common neurological disease published in December 2023?”
docs = db.similarity_search(query)
docs
[Document(page_content=”Neurological disorders constitute a significant
portion of the global disease burden, affecting >30% of the world’s population.
This prevalence poses a substantial threat to global health in the foreseeable
future. A lack of awareness regarding this high burden of neurological
diseases has led to their underrecognition, underappreciation, and
insufficient funding. Establishing a strategic and comprehensive research
agenda for brain-related studies is a crucial step towards aligning research
objectives among all pertinent stakeholders and fostering greater societal
awareness.”, metadata={‘day’: ’12’, ‘month’: ’12’, ‘seq_num’: 6930,
‘source’: ‘./pubmed_article_december-2023.json’, ‘title’: ‘A strategic
neurological research agenda for Europe: Towards clinically relevant and
patient-centred neurological research priorities.’, ‘year’: ‘2023’}),
Document(page_content=’ frame of future research endeavors against
neurodegenerative/neural diseases have also been briefly discussed.’,
metadata={‘day’: ’16’, ‘month’: ’12’, ‘seq_num’: 599,
‘source’: ‘./pubmed_article_december-2023.json’, ‘title’: ‘Neuroprotective
Action of Humanin and Humanin Analogues: Research Findings and Perspectives.’,
‘year’: ‘2023’}), Document(page_content=’ the mid-21st century, especially
under the unrestricted-emission scenario. These results highlight the urgent
need for effective climate and public health policies to address the growing
challenges of neurodegenerative diseases associated with global warming.’,
metadata={‘day’: ’12’, ‘month’: ’12’, ‘seq_num’: 6660,
‘source’: ‘./pubmed_article_december-2023.json’, ‘title’: ‘Temperature-related
death burden of various neurodegenerative diseases under climate warming: a
nationwide modelling study.’, ‘year’: ‘2023’})]
从结果中,成功地检索了 2 个与查询相关的块。
加载预训练 LLM
我们将从 HuggingFace 加载预先训练的 Mistral 7B。第一步涉及登录 HuggingFace 中心。请注意,你需要使用 HF API Token 来执行此操作:
import os
from huggingface_hub import notebook_login
os.environ[‘HUGGINGFACEHUB_API_TOKEN’]=’YOUR_HF_API_TOKEN’
notebook_login()After this, let’s proceed with loading Mistral 7B:
之后,加载 Mistral 7B:
from transformers import AutoTokenizer, AutoModelForCausalLM
from transformers import AutoTokenizer, pipeline
from langchain import HuggingFacePipeline
model_id = “mistralai/Mistral-7B-v0.1”
tokenizer = AutoTokenizer.from_pretrained(model_id)
model = AutoModelForCausalLM.from_pretrained(model_id, load_in_8bit=False, device_map=’auto’)
pipe = pipeline(“text-generation”, model=model, tokenizer=tokenizer, max_new_tokens=128)
llm = HuggingFacePipeline(
pipeline = pipe,
model_kwargs={“temperature”: 0.5, “max_length”: 512}
)
构建 RAG 管道
设置好 LLM 和向量数据库后,我们现在可以使用自定义的提示模板定义 LangChain 管道:
# Define the langchain pipeline
from langchain.chains import RetrievalQA
from langchain.prompts import PromptTemplate
import time
PROMPT_TEMPLATE = “””Answer the question based only on the following context:
{context}
You are allowed to rephrase the answer based on the context.
Question: {question}
“””
PROMPT = PromptTemplate.from_template(PROMPT_TEMPLATE)
qa_chain = RetrievalQA.from_chain_type(
llm,
retriever=db.as_retriever(k=2),
chain_type_kwargs={“prompt”: PROMPT},
return_source_documents=True
)
现在,我们将通过以下测试来评估我们的 LLM+RAG 组合系统:
- 一个问题
- 执行时间
- 生成响应时使用的检索到相关联的元数据。
start_time = time.time()
#Query
query = “What are the safest cryopreservation methods?”
result = qa_chain({“query”: query})
#Execution Time
print(f”\n— {time.time() – start_time} seconds —“)
print(result[‘result’].strip())
#Metadata
titles = [‘\t-‘+doc.metadata[‘title’] for doc in result[‘source_documents’]]
print(“\n\nThe provided answer is based on the following PubMed articles:\t”)
print(“\n”.join(set(titles)))
结果:
— 6.252970457077026 seconds —
The safest cryopreservation methods are those that use ionic liquids,
deep eutectic solvents, or certain polymers, which open the door to new
cryopreservation methods and are also less toxic to frozen samples.
The provided answer is based on the following PubMed articles:
-Advances in Cryopreservatives: Exploring Safer Alternatives.
-In Vitro and In Silico Antioxidant Activity and Molecular Characterization of Bauhinia ungulata Essential Oil.
-A new apotirucallane-type protolimonoid from the leaves of <i>Paramignya trimera</i>.
现在,我们将继续仅使用 LLM 测试:
# Define the langchain pipeline for llm only
from langchain.prompts import PromptTemplate
PROMPT_TEMPLATE =”””Answer the given Question only. Your answer should be in your own words and be no longer than 100 words. \n\n Question: {question} \n\n
Answer:
“””
PROMPT = PromptTemplate.from_template(PROMPT_TEMPLATE)
llm_chain = PROMPT | llm
start_time = time.time()
result = llm_chain.invoke({“question”: query})
print(f”\n— {time.time() – start_time} seconds —“)
print(result[‘result’].strip())
结果:
— 6.095802927251892 seconds —
I’m not sure what you mean by safest. If you mean safest for the patient,
then I would say that the safest method is to not be cryopreserved at all.
比较这两个结果,很明显,LLM+RAG 组合比单独使用 LLM 产生更准确和详细的答案。此外,RAG 还具有透明度的优势。通过引用 PubMed 来源,用户可以验证答案的准确性,从而增加对模型答案的信任。然而,值得注意的是,这些改进会带来计算成本。与仅使用 LLM 相比,同时使用检索和 LLM 的系统的执行时间要慢。
总结
通过本文的研究,检索增强生成 (RAG) 与 Mistral 7B 等大型语言模型 (LLM) 的集成显著提高了医学问答中答案的准确性和可靠性。这项研究使用最新的 MEDLINE 数据库数据,强调了 RAG 在处理复杂的医学查询方面改进 LLM 的能力。 RAG 增强方法不仅提供更准确的响应,而且还通过源引用增加了一层透明度。
未来的应用,例如将 RAG-LLM 管道嵌入到聊天机器人界面中,使用户能够直接与前沿研究成果进行交互。
本文翻译 Build a Medical Q&A system using LangChain and Mistral 7B。










暂无评论内容